 |
| Na foto, Jane Goodall, pioneira e uma das maiores especialistas do mundo no estudo de comportamento social em primatas. Imagem disponível aqui.
O texto anterior desta série (que pode ser lido aqui) trouxe ideias acerca de usar o DNA dos organismos para organizá-los sob uma perspectiva evolutiva, utilizando a sistemática. Aproveitando esse gancho, neste texto faremos algo análogo ao texto 3, um exercício de reconstruir a evolução de um grupo muito interessante (para nós, pelo menos). Porém, desta vez, ao invés de usarmos características físicas dos organismos, faremos uso de sequências de "letras" do DNA.
|
Da mesma forma que fiz no texto 3, é bom deixar claro que, esse trabalho de dois dias é raso e tratará apenas de um fragmento do DNA dos organismos, e não deve ser visto com o rigor de uma análise real. Para termos uma ideia, o trabalho de Chiari e colaboradores sobre posicionamento das tartarugas (grupo Testudines) dentro dos répteis, que já foi citado em um texto anterior do blog (para lê-lo clique aqui), utilizou 1000 marcadores genéticos diferentes e demandou o trabalho de meses e muitos profissionais da área. [1]
GRUPO DE INTERESSE
É muito comum ouvirmos falar sobre nós humanos compartilharmos quase 100% do DNA, em termos de sequências de bases, com os Chimpanzés. O que isso quer dizer, em termos cladisticos, é que a cladogenese que separou a população ancestral que teria dado origem à nossa linhagem e à dos nossos primos do gêneros Pan é muito recente. Poucos milhões de anos atrás, essa população, ainda existia como especie única, e esse pouco tempo de separação, explica uma diferença genética muito pequena nas sequências de DNA entre nós e nossos primos mais próximos vivos.
Hoje tentaremos reconstruir essa hipótese filogenética, em uma rápida análise de como foi a evolução dos primatas, grupo que surgiu à menos de 100 milhões de anos atrás, e confirmar se a ideia do parágrafo acima realmente se confirma na prática.
OBTENDO SEQUÊNCIAS
Há duas formas de obter sequências genéticas: a primeira seria sequenciando nós mesmos o DNA dos organismos. Isso envolveria muito dinheiro, o sequestro de vários primatas do zoológico mais próximo e técnicas laboratoriais de sequenciamento genético.
Como a primeira, além de infringir leis, está além das minhas condições financeiras, vamos recorrer a uma forma alternativa. Para praticamente todos os trabalhos publicados no qual se fazem sequenciamento genético, os dados obtidos são publicados em bancos de dados abertos, como por exemplo o Genbank [2]. Isso garante a transparência e replicabilidade das pesquisas (afinal, estamos falando de ciência). E é nesses bancos de dados que poderemos obter, de graça, as nossas sequências para o exercício.
ESCOLHA DOS GENES
Para uma análise molecular podemos usar desde uma sequência curta, de um único gene de 800 bases, até o genoma completo dos organismos, que pode ser da ordem de bilhões de nucleotídeos. É claro que, quanto maior a sequência, mais trabalho nosso computador vai ter. Como o meu já está desafiando a obsolescência programada com seus 7 anos de idade, nós usaremos os genomas mitocondriais dos organismos, um sequenciamento popular e com boa quantidade de informação filogenética.
GENOMA MICONDRIAL?
Dentro das células da maioria dos eucariotos (aqueles que possuem células com núcleo), existem pequenas organelas responsáveis pela maquinaria bioquímica de produção de energia dos seres vivos, chamadas de mitocôndrias (há uma história fantástica acerca de como essa organela foi parar lá, mas essa história foge ao escopo do texto atual, talvez um dia ela seja contada aqui no blog).[3]
 |
| Imagem disponível aqui. |
Dentro das mitocôndrias existe um genoma circular, na verdade cada organela pode conter de 2 a 10 cópias dele, contendo 22 genes que codificam RNA transportador, duas regiões que codificam RNA ribossômico e 13 proteínas. Essa informação toda esta organizada em um conjunto de cerca de 16500 pares de bases nitrogenadas e é o que garante a transformação da glicose dos alimentos em energia útil para todos os processos que o organismo realiza. [3][4]
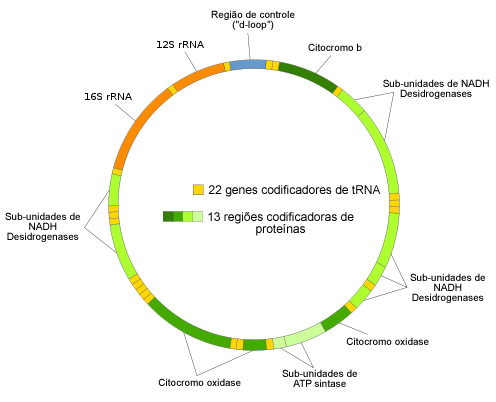 |
| Imagem disponível aqui. |
É essa sequência de bases que será usada para a nossa análise. Selecionamos 63 sequências de primatas de todas as 16 famílias da ordem (cerca de 15% de todas as 429 espécies válidas, sendo a terceira maior ordem de mamíferos viventes, perdendo apenas para roedores e morcegos ), deste total, 50 gêneros estão representados na análise, mais de 60% do total de gêneros válidos.[5]
Também faremos uma polarização, como no exercício do texto 3, mas agora ao invés dos peixes Dipnoicos, usaremos como grupo externo uma ordem de mamíferos próxima evolutivamente dos primatas: os Colugos (ordem Dermoptera). Também conhecidos como lêmures voadores, a ordem possui apenas duas espécies viventes, habitando as árvores do sudeste asiático. Estes animais usam uma membrana que liga o corpo aos membros para planar entre árvores (daí o nome popular lêmure voador).[6]
 |
| Imagem aqui. |
ALINHAMENTO DE SEQUÊNCIAS
Depois de baixar as sequências, precisamos alinhá-las, isso é necessário pois cada organismo possui um genoma mitocondrial de determinado tamanho (mutações no DNA de uma linhagem podem alterar o tamanho das sequências).
O processo de alinhamento insere "gaps" (buracos) dentro das sequências para buscar um alinhamento perfeito afim de conseguir alinhar os trechos homólogos. Esse é o primeiro desafio computacional: buscar entre milhões de formas de alinhar as sequências, um alinhamento ideal. Esse processo é ainda mais custoso que a busca por árvores parcimoniosas, e também requer mecanismos heurísticos (para saber mais leia o texto 4).[7]
Usando um software de alinhamento (eu usei o Bioedit), fazemos o alinhamento das nossas sequências. Vejam na figura abaixo como os dados começaram a se arranjar melhor após esse procedimento que levou cerca de 12 horas.
ANÁLISE CLADÍSTICA
Após a limpeza do alinhamento (removendo as pontas e deixando só as regiões de interesse) montamos a matriz para buscarmos pela árvore mais parcimoniosa.
OBS: Para dados moleculares há formas alternativas para buscar pela melhor árvore, além do critério de máxima parcimônia. Existem métodos que levam em conta modelos de substituição de bases no DNA com diferentes custos pra cada mudança nos nucleotídeos, como os algoritmos de Maximum Likelihood. Há ainda modelos Bayesianos que inserem o componente aleatório das mutações nas análises.
Para a análise da nossa matriz, vamos usar o programa TNT. Abrimos o TNT, selecionamos a entrada de dados como DNA. Abriremos nossa matriz e, a partir dela, o programa buscará pela árvore que reconstrói a evolução das sequências, de forma a usar o menor número de mudanças possíveis.
Após o fim da análise chegamos a uma única árvore.
 | |
|
Podemos ver na nossa reconstrução diversos grupos monofiléticos. Cada cor nos ramos representa uma família de primatas. Nossa análise mostra que todas as 16 famílias tradicionais, exceto uma (a família Lorisidae) é monofilética. Obviamente, a nossa análise não nos permite afirmar que essa família não é um grupo natural; para tanto, seria necessário muito mais rigor em uma análise muito mais robusta que a nossa. As cores por fora da arvore representam as classificações mais abrangentes, também monofiléticas, dentro do grupo dos primatas.
Grupos como o dos primatas do velho mundo, chamados cientificamente de Catharrhiny ( representado pela junção dos clados Cercophitecoidea e Homininoidea) e dos primatas do novo mundo (grupo Platyrrhini) também foram recuperados. Não está no escopo desse texto falar sobre a evolução dos primatas a fundo (isso será abordado futuramente, em outra série de textos).
Por questões didáticas e para economizar o tempo dos interessados por conhecer a carinha dos primatas de cada grupo segue umas fotos e um resumo de cada um deles:
Tarsiiformes: é o grupo dos tarsos e primatas relacionados, sua posição filogenética hoje é reconhecida e difere do resultado da nossa análise, sendo eles mais próximos dos simiiformes que dos pro-símios como lêmures.

Por questões didáticas e para economizar o tempo dos interessados por conhecer a carinha dos primatas de cada grupo segue umas fotos e um resumo de cada um deles:
Tarsiiformes: é o grupo dos tarsos e primatas relacionados, sua posição filogenética hoje é reconhecida e difere do resultado da nossa análise, sendo eles mais próximos dos simiiformes que dos pro-símios como lêmures.

Lorisoidea: o grupo que compreende os loris e galagos. A posição obtida pela nossa analise é coerente com as hipóteses atuais.
Lemuroidea: grupos inclui todos os lemuriformes e sua posição tanto em nossa análise quanto nas hipóteses atuais é corroborada como grupo irmão de Lorisoidea.

Platyrrhini: compreende todos os macacos do novo mundo, e as principais espécies brasileiras como o macaco-prego, macaco-aranha e mico leão-dourado. Sua posição filogenética é sólida como grupo irmão dos macacos do velho mundo ou Catharrhini.
Cercophitecoidea: são grupo irmão da linhagem Hominoidea, que juntos formam o grupo dos macacos do velho mundo. São todos macacos nativos do oriente na África e na Ásia. Alguns integrantes bastante conhecidos são os babuínos, macacos rhesus e o mandril.
E agora, o que mais nos interessa nesse exercício: a pequena família de apenas 7 espécies viventes, na qual estamos incluídos: a família Hominidae e seu grupo irmão Hylobatidae (os gibões). Vamos dar um zoom nestas famílias e ver o que nossa análise nos mostra.
Como podemos observar, a hipótese mais aceita atualmente foi obtida. O primeiro grupo (de hominídeos) a se diversificar foi a linhagem dos Ponginae, depois temos os gorilas como irmãos de um grupo formado por nós e as duas espécies do gênero Pan, o Chimpanzé comum (Pan troglodytes) e os Bonobos (Pan paniscus).
Com essa breve análise fica evidente que há um ancestral comum recente entre nós e os macacos de forma geral, e mais intimamente com os Chimpanzés. Esse posicionamento é corroborado por inúmeros trabalhos tanto morfológicos quanto moleculares muito mais robustos, incluindo uma filogenia molecular com 54 genes, totalizando uma matriz de 35000 nucleotídeos.[8][9]
CONCLUSÃO
Nosso DNA, dados morfológicos e evidências fósseis deixam claro um fato incontestável, que mesmo que questionado por muitos negacionistas, continua sendo científicamente aceito e já muito bem assentado na academia científica: somos sim irmãos dos Chimpanzés e de tantos outros primatas. Nós somos, incontestavelmente, macacos.
No próximo texto desta série veremos de que forma os fósseis são tratados na sistemática, como eles são essenciais para resolver problemas sobre a evolução dos grupos e também algumas confusões que podem ocorrer ao omiti-los das filogenias, até o próximo texto!
O texto 6, sobre fósseis e sistematica já está disponível no blog, basta clicar aqui e continuar a leitura.
O texto 6, sobre fósseis e sistematica já está disponível no blog, basta clicar aqui e continuar a leitura.
REFERÊNCIAS
1 - Chiari, Ylenia, et al. "Phylogenomic analyses support the position of turtles as the sister group of birds and crocodiles (Archosauria)." Bmc Biology 10.1 (2012): 65.
2 -Benson, Dennis A., et al. "GenBank." Nucleic acids research36.Database issue (2008): D25.
3 - Siekevitz P (1957). "Powerhouse of the cell". Scientific American. 197 131-140
4 - Anderson, S.; Bankier, A. T.; Barrell, B. G.; de Bruijn, M. H. L.; Coulson, A. R.; Drouin, J.; Eperon, I. C.; Nierlich, D. P.; Roe, B. A.; Sanger, F.; Schreier, P. H.; Smith, A. J. H.; Staden, R.; Young, I. G. (1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–65
5 - List of primates. Encyclopædia Britannica, Abril 07, 2016. Disponível em:https://www.britannica.com/topic/list-of-primates-2060305
6 - Schmitz, Jürgen, et al. "The colugo (Cynocephalus variegatus, Dermoptera): the primates' gliding sister?." Molecular Biology and Evolution 19.12 (2002): 2308-2312.
7-DeSalle, Rob, Gonzalo Giribet, and Ward Wheeler, eds. Multiple sequence alignment, Molecular Systematics and Evolution: theory and practice.
8-Perelman, Polina, et al. "A molecular phylogeny of living primates." PLoS genetics 7.3 (2011): e1001342.
9-Nengo, Isaiah, et al. "New infant cranium from the African Miocene sheds light on ape evolution." Nature 548.7666 (2017): 169.











Nenhum comentário:
Postar um comentário